Chem. Sci. | 陈红明课题组与中山大学团队合作提出基于分子片段的人工智能分子设计新算法
来源: 本网 作者: 本网 日期: 2020-08-09
导读:
近日英国皇家化学会核心期刊Chemical Science在线刊登了生物岛实验室的陈红明研究员与中山大学药学院药物分子设计中心徐峻教授的联合研究成果,“SyntaLinker: automatic fragment linking with deep conditional transformer neural networks“。他们在文章中提出了一种新的基于带约束Transformer神经网络的分子设计方法,这种方法可以利用神经网络自动连接两个分子碎片,设计满足一定链接段(Linker)约束条件的小分子分子结构。

近年来,深度学习技术在人工智能研究领域取得了巨大突破。其中基于深度学习的生成模型算法,结合传统的药物分子设计理念,在药学领域引发了学习的浪潮。深度学习在不依赖任何人为预定义规则的情况下,通过数据驱动下的自主学习,能快速自动生成大批结构类似的类药性分子。当前,传统的生成模型大都是基于整个化合物分子的生成方式,这种生成模型算法在一定程度上降低了生成分子结构的多样性和新颖度,而基于片段的生成算法则有希望解决这一问题。此外,在基于片段的药物设计中,片段连接一直是具有很大挑战性的问题,如何生成满足一定链接段(Linker)约束条件,使得被链接的终端片段能够保持原有的结合模式,同时又满足类药性条件,且结构多样的分子是迫切需要解决的问题。
陈红明研究员与中山大学药学院药物分子设计中心徐峻教授研究团队,联合提出了带约束的Transformer神经网络架构SyntaLinker,将待连接的终端片段的最短连接段键长及部分药效团属性作为神经网络的约束条件,可以快速自动生成满足特定链接段约束条件的大量新颖的分子结构。这种带约束的Transformer 神经网络SyntaLinker本质上是由多个注意力机制(Attention)模块构成的,SyntaLinker利用其编码层和解码层对输入的分子片段结构序列进行处理,将分子片段自动连接起来,并且结合约束信息,填充链接段,从而生成一个完整的分子。在模型训练过程中,通过对来源于ChEMBL数据库的大量化学结构进行分析,学习其中包含的药物分子片段组装的隐式规则。而在片段连接即新分子设计阶段,只需要提供两端的终端片段结构和人为定义的链接段约束条件,SyntaLinker就可以产生大量满足约束条件的连接后的分子结构。研究中,研究团队尝试用此算法对多个药物设计问题进行了验证,包括:先导化合物优化、片段连接、骨架跃迁。SyntaLinker模型不仅能复现出真实实验过程中的实验优化及片段连接案例,还获得了一批额外的符合预期的分子,并在计算水平上验证了分子的优良性(具有更高的靶标蛋白对接打分值和较高的3D分子相似度)。我们期待未来这种基于片段连接的分子设计算法能被用于实际的药物开发项目中,为药物化学家提供更多带启发性的化学结构。
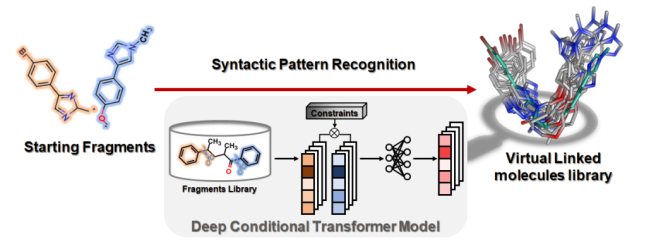
带约束Transformer神经网络(SyntaLinker)架构
陈红明研究员简介:
陈红明研究员,先后在德国拜耳公司和阿斯利康制药公司任职,从事计算化学和新药开发工作长达20年,曾在阿斯利康制药公司瑞典哥德堡研发中心先导化合物发现部门担任主任研究员(Principal Scientist)职位。2019年加盟生物岛实验室,组建人工智能与药物设计研发团队。主要研究兴趣集中在计算化学,化学信息学,人工智能/机器学习等方面,已发表学术论文和专利70余篇。担任Molecular Informatics 杂志学术咨询委员会委员。